参考 Delta Lake まずはjpgをSparkで読み込む 予め、Delta Lakeを読み込みながら起動する。
1 $ /opt/spark/default/bin/spark-shell --packages io.delta:delta-core_2.11:0.6.0
Spark公式ドキュメントのImage
data source を利用し、jpgをDataFrameとして読み込んでみる。
データは、 Kaggleのmnistデータ
を利用。このデータはjpgになっているので便利。
1 2 3 scala> val home = sys.env("HOME" ) scala> val imageDir = "Downloads/mnist_jpg/trainingSet/trainingSet/0" scala> val df = spark.read.format("image" ).option("dropInvalid" , true ).load(home + "/" + imageDir)
データをDataFrameにする定義ができた。 内容は以下の通り。
1 2 3 4 5 6 7 8 scala> df.select("image.origin" , "image.width" , "image.height" ).show(3 , truncate=false ) +-------------------------------------------------------------------------------+-----+------+ |origin |width|height| +-------------------------------------------------------------------------------+-----+------+ |file: |file: |file: +-------------------------------------------------------------------------------+-----+------+
公式ドキュメントのとおりだが、カラムは以下の通り。
origin: StringType (represents the file path of the image)
height: IntegerType (height of the image)
width: IntegerType (width of the image)
nChannels: IntegerType (number of image channels)
mode: IntegerType (OpenCV-compatible type)
data: BinaryType (Image bytes in OpenCV-compatible order: row-wise
BGR in most cases)
1 2 3 4 5 6 7 8 9 scala> df.printSchema root |-- image: struct (nullable = true ) | |-- origin: string (nullable = true ) | |-- height: integer (nullable = true ) | |-- width: integer (nullable = true ) | |-- nChannels: integer (nullable = true ) | |-- mode: integer (nullable = true ) | |-- data: binary (nullable = true )
Delta Lakeで扱う 書き込み これをDelta LakeのData Sourceで書き出してみる。
1 2 scala> val deltaImagePath = "/tmp/delta-lake-image" scala> df.write.format("delta" ).save(deltaImagePath)
読み出し できた。試しにDeltaTableとして読み出してみる。
1 2 3 scala> import io.delta.tables._ scala> import org.apache.spark.sql.functions._ scala> val deltaTable = DeltaTable .forPath(deltaImagePath)
更新 mergeを試す。
マージのためにキーを明示的に与えつつデータを準備する。
1 2 3 4 5 scala> val targetDf = df.select($"image.origin" , $"image" ) scala> val targetImagePath = "/tmp/delta-table" scala> targetDf.write.format("delta" ).save(targetImagePath) scala> val sourcePath = "Downloads/mnist_jpg/trainingSet/trainingSet/1" scala> val sourceDf = spark.read.format("image" ).option("dropInvalid" , true ).load(home + "/" + sourcePath).select($"image.origin" , $"image" )
Delta Tableを定義。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 scala> val deltaTable = DeltaTable .forPath(targetImagePath) scala> deltaTable .as("target" ) .merge( sourceDf.as("source" ), "target.origin = source.origin" ) .whenMatched .updateExpr(Map ( "image" -> "source.image" )) .whenNotMatched .insertExpr(Map ( "origin" -> "source.origin" , "image" -> "source.image" )) .execute()
実際に、追加データが入っていることが確かめられる。
1 2 3 4 5 6 7 8 9 scala> deltaTable.toDF.where($"origin" contains "trainingSet/1" ).select("image.origin" , "image.width" , "image.height" ).show(3 , truncate=false ) +-------------------------------------------------------------------------------+-----+------+ |origin |width|height| +-------------------------------------------------------------------------------+-----+------+ |file: |file: |file: +-------------------------------------------------------------------------------+-----+------+ only showing top 3 rows
SparkのImage Data
Sourceは、メタデータと画像本体(バイナリ)を構造で扱っているだけなので、
実は特に工夫することなく扱えた。
公式ドキュメントによると、OpenCV形式バイナリで保持されている。
これから先は、Python使って処理したいので、改めて開き直す。
Parquetの内容を確認する parquet-mr の
parquet-tools を利用して、Parquetの内容を確認する。
なお、Hudi
v0.5.2におけるParquetのバージョンは以下の通り、1.10.1である。
pom.xml:84
1 <parquet.version > 1.10.1</parquet.version >
当該バージョンに対応するタグをチェックアウトし、予めparquet-toolsをパッケージしておくこと。
スキーマ 1 2 3 4 5 6 7 8 9 10 11 $ java -jar target/parquet-tools-1.10.1.jar schema /tmp/delta-lake-image/part-00000-c3d03d70-1785-435f-b055-b2a78903e732-c000.snappy.parquet message spark_schema { optional group image { optional binary origin (UTF8); optional int32 height; optional int32 width; optional int32 nChannels; optional int32 mode; optional binary data; } }
シンプルにParquet内に保持されていることがわかる。 Spark
SQLのParquet取扱機能を利用している。
メタデータ ファイル名やクリエイタなど。
1 2 3 file: file:/tmp/delta-lake-image/part-00000-c3d03d70-1785-435f-b055-b2a78903e732-c000.snappy.parquet creator: parquet-mr version 1.10.1 (build a89df8f9932b6ef6633d06069e50c9b7970bebd1) extra: org.apache.spark.sql.parquet.row.metadata = {"type":"struct","fields":[{"name":"image","type":{"type":"struct","fields":[{"name":"origin","type":"string","nullable":true,"metadata":{}},{"name":"height","type":"integer","nullable":true,"metadata":{}},{"name":"width","type":"integer","nullable":true,"metadata":{}},{"name":"nChannels","type":"integer","nullable":true,"metadata":{}},{"name":"mode","type":"integer","nullable":true,"metadata":{}},{"name":"data","type":"binary","nullable":true,"metadata":{}}]},"nullable":true,"metadata":{}}]}
ファイルスキーマ
1 2 3 4 5 6 7 8 9 file schema: spark_schema -------------------------------------------------------------------------------- image: OPTIONAL F:6 .origin: OPTIONAL BINARY O:UTF8 R:0 D:2 .height: OPTIONAL INT32 R:0 D:2 .width: OPTIONAL INT32 R:0 D:2 .nChannels: OPTIONAL INT32 R:0 D:2 .mode: OPTIONAL INT32 R:0 D:2 .data: OPTIONAL BINARY R:0 D:2
Rowグループ
1 2 3 4 5 6 7 8 9 10 11 row group 1: RC:32 TS:29939 OFFSET:4 -------------------------------------------------------------------------------- image: .origin: BINARY SNAPPY DO:0 FPO:4 SZ:620/2838/4.58 VC:32 ENC:RLE,PLAIN,BIT_PACKED ST:[min: file:///home/centos/Downloads/mnist_jpg/trainingSet/trainingSet/0/img_10203.jpg, max: file:///home/centos/Downloads/mnist_jpg/trainingSet/trainingSet/0/img_9098.jpg, num_nulls: 0] .height: INT32 SNAPPY DO:0 FPO:624 SZ:74/70/0.95 VC:32 ENC:RLE,PLAIN_DICTIONARY,BIT_PACKED ST:[min: 28, max: 28, num_nulls: 0] .width: INT32 SNAPPY DO:0 FPO:698 SZ:74/70/0.95 VC:32 ENC:RLE,PLAIN_DICTIONARY,BIT_PACKED ST:[min: 28, max: 28, num_nulls: 0] .nChannels: INT32 SNAPPY DO:0 FPO:772 SZ:74/70/0.95 VC:32 ENC:RLE,PLAIN_DICTIONARY,BIT_PACKED ST:[min: 1, max: 1, num_nulls: 0] .mode: INT32 SNAPPY DO:0 FPO:846 SZ:74/70/0.95 VC:32 ENC:RLE,PLAIN_DICTIONARY,BIT_PACKED ST:[min: 0, max: 0, num_nulls: 0] .data: BINARY SNAPPY DO:0 FPO:920 SZ:21175/26821/1.27 VC:32 ENC:RLE,PLAIN,BIT_PACKED ST:[min: 0x00 (snip)
Pythonで画像処理してみる とりあえずOpenCVをインストールしたPython環境のJupyterでPySparkを起動。
1 $ PYSPARK_DRIVER_PYTHON_OPTS="notebook --ip 0.0.0.0" /opt/spark/default/bin/pyspark --packages io.delta:delta-core_2.11:0.6.0 --conf spark.pyspark.driver.python=$HOME /venv/jupyter/bin/jupyter
この後はJupyter内で処理する。
1 2 3 4 5 from delta.tables import *from pyspark.sql.functions import *delta_image_path = "/tmp/delta-lake-image" deltaTable = DeltaTable.forPath(spark, delta_image_path) deltaTable.toDF().select("image.origin" , "image.width" , "image.height" ).show(3 , truncate=False )
とりあえず読み込めたことがわかる。
1 2 3 4 5 6 7 8 +-------------------------------------------------------------------------------+-----+------+ |origin |width|height| +-------------------------------------------------------------------------------+-----+------+ |file:///home/centos/Downloads/mnist_jpg/trainingSet/trainingSet/0/img_20188.jpg|28 |28 | |file:///home/centos/Downloads/mnist_jpg/trainingSet/trainingSet/0/img_12634.jpg|28 |28 | |file:///home/centos/Downloads/mnist_jpg/trainingSet/trainingSet/0/img_26812.jpg|28 |28 | +-------------------------------------------------------------------------------+-----+------+ only showing top 3 rows
中のデータを利用できることを確かめるため、1件だけ取り出して処理してみる。
1 img = deltaTable.toDF().where("image.origin == 'file:///home/centos/Downloads/mnist_jpg/trainingSet/trainingSet/0/img_1.jpg'" ).select("image.origin" , "image.width" , "image.height" , "image.nChannels" , "image.data" ).collect()[0 ]
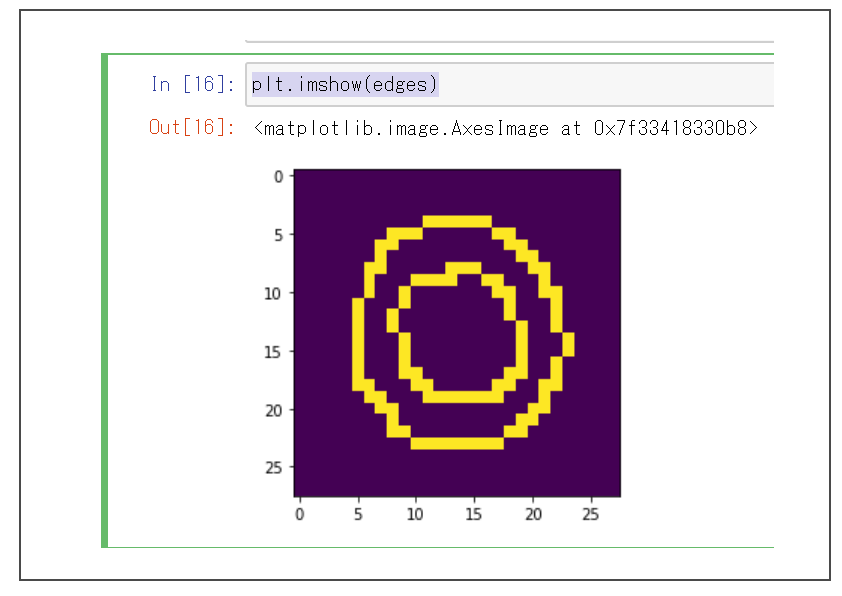
とりあえずエッジ抽出をしてみる。
1 2 3 4 5 6 7 import numpy as npimport cv2from matplotlib import pyplot as pltnparr = np.frombuffer(img.data, np.uint8).reshape(img.height, img.width, img.nChannels) edges = cv2.Canny(nparr,100 ,200 ) plt.imshow(edges)
Jupyter上でエッジ抽出されたことが確かめられただろうか。
エッジ抽出された様
これをSparkでUDFで実行するようにする。
(とりあえず雑にライブラリをインポート…)
1 2 3 4 5 6 7 8 9 10 11 def get_edge (data, h, w, c) : import numpy as np import cv2 nparr = np.frombuffer(data, np.uint8).reshape(h, w, c) edge = cv2.Canny(nparr,100 ,200 ) return edge.tobytes() from pyspark.sql.functions import udffrom pyspark.sql.types import BinaryTypeget_edge_udf = udf(get_edge, BinaryType())
エッジ抽出されたndarrayをバイト列に変換し、Spark上ではBinaryTypeで扱うように指定した。
1 2 3 with_edge = deltaTable.toDF().select("image.origin" , "image.width" , "image.height" , "image.nChannels" , get_edge_udf("image.data" , "image.height" , "image.width" , "image.nChannels" ).alias("edge" ), "image.data" ) with_edge.show(3 )
1 2 3 4 5 6 7 8 +--------------------+-----+------+---------+--------------------+--------------------+ | origin|width|height|nChannels| edge| data| +--------------------+-----+------+---------+--------------------+--------------------+ |file:///home/cent...| 28 | 28 | 1 |[00 00 00 00 00 0. ..|[05 00 04 08 00 0. ..| |file:///home/cent...| 28 | 28 | 1 |[00 00 00 00 00 0. ..|[05 00 0 B 00 00 0. ..| |file:///home/cent...| 28 | 28 | 1 |[00 00 00 00 00 0. ..|[06 00 02 00 02 0. ..| +--------------------+-----+------+---------+--------------------+--------------------+ only showing top 3 rows
こんな感じで、OpenCVを用いた処理をDataFrameに対して実行できる。
なお、当然ながら処理されたデータを取り出せば、また画像として表示も可能。
1 2 3 new_img = with_edge.where("origin == 'file:///home/centos/Downloads/mnist_jpg/trainingSet/trainingSet/0/img_1.jpg'" ).select("origin" , "width" , "height" , "nChannels" , "data" , "edge" ).collect()[0 ] new_nparr = np.frombuffer(new_img.edge, np.uint8).reshape(new_img.height, new_img.width) plt.imshow(new_nparr)
Hudi 画像読み込み まずはjpgをSparkで読み込む
と同様に データを読み込む。
Hudiのquick-start-guide
を参考にしながら、まずはHudiを読み込みながら、シェルを起動する。
1 2 $ /opt/spark/default/bin/spark-shell --packages org.apache.hudi:hudi-spark-bundle_2.11:0.5.1-incubating,org.apache.spark:spark-avro_2.11:2.4.5 \ --conf 'spark.serializer=org.apache.spark.serializer.KryoSerializer'
ライブラリをインポート。
1 2 3 4 scala> import org.apache.spark.sql.SaveMode ._ scala> import org.apache.hudi.DataSourceReadOptions ._ scala> import org.apache.hudi.DataSourceWriteOptions ._ scala> import org.apache.hudi.config.HoodieWriteConfig ._
画像データを読み込み
1 2 3 4 5 scala> val tableName = "hudi_images" scala> val basePath = "file:///tmp/hudi_images" scala> val home = sys.env("HOME" ) scala> val imageDir = "Downloads/mnist_jpg/trainingSet/trainingSet/*" scala> val df = spark.read.format("image" ).option("dropInvalid" , true ).load(home + "/" + imageDir)
今回は、Hudiにおけるパーティション構成を確認するため、上記のようにワイルドカード指定した。
書き込み Hudi書き込み用にデータを変換。パーティション用キーなどを定義する。
1 2 3 4 5 scala> import org.apache.spark.sql.functions.{col, udf} scala> val partitionPath = udf((str: String ) => { str.split("/" ).takeRight(2 ).head }) scala> val hudiDf = df.select($"image.origin" , partitionPath($"image.origin" ) as "partitionpath" , $"*" )
書き込み。
1 2 3 4 5 6 7 scala> hudiDf.write.format("hudi" ). option(TABLE_NAME , tableName). option(PRECOMBINE_FIELD_OPT_KEY , "origin" ). option(RECORDKEY_FIELD_OPT_KEY , "origin" ). option(PARTITIONPATH_FIELD_OPT_KEY , "partitionpath" ). mode(Overwrite ). save(basePath)
書き込まれたデータは以下のような感じ。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 $ ls -R /tmp/hudi_images/ /tmp/hudi_images/: 0 1 2 3 4 5 6 7 8 9 /tmp/hudi_images/0: 86c74288-e541-4a25-addf-0c00e17ef6bf-0_0-29-14074_20200517135104.parquet /tmp/hudi_images/1: 4912032f-3365-4852-b2ae-91ffb5ea9806-0_1-29-14075_20200517135104.parquet /tmp/hudi_images/2: 0636bb93-dd32-40f1-b5c8-328130386528-0_2-29-14076_20200517135104.parquet /tmp/hudi_images/3: 6f0154cd-4a9f-4001-935b-47d067416797-0_3-29-14077_20200517135104.parquet /tmp/hudi_images/4: b6f29c2c-7df0-481a-8149-2bbc93e92e2c-0_4-29-14078_20200517135104.parquet /tmp/hudi_images/5: 24115172-10db-4c85-808a-bf4048e0f533-0_5-29-14079_20200517135104.parquet /tmp/hudi_images/6: 64823f4b-26fb-4679-87e8-cd81d01b1181-0_6-29-14080_20200517135104.parquet /tmp/hudi_images/7: d0c097e3-9ab7-477a-b591-593c6cb7880f-0_7-29-14081_20200517135104.parquet /tmp/hudi_images/8: 13a48b38-cbfb-4076-957d-9c580765bfca-0_8-29-14082_20200517135104.parquet /tmp/hudi_images/9: 5d3ba9ae-8ede-410f-95f3-e8d69e8c0478-0_9-29-14083_20200517135104.parquet
メタデータは以下のような感じ。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 $ ls -1 /tmp/hudi_images/.hoodie/ 20200517135104.clean 20200517135104.clean.inflight 20200517135104.clean.requested 20200517135104.commit 20200517135104.commit.requested 20200517135104.inflight 20200517140006.clean 20200517140006.clean.inflight 20200517140006.clean.requested 20200517140006.commit 20200517140006.commit.requested 20200517140006.inflight archived hoodie.properties
読み込み 読み込んで見る。
1 2 3 4 scala> val hudiImageDf = spark. read. format("hudi" ). load(basePath + "/*" )
スキーマは以下の通り。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 scala> hudiImageDf.printSchema root |-- _hoodie_commit_time: string (nullable = true ) |-- _hoodie_commit_seqno: string (nullable = true ) |-- _hoodie_record_key: string (nullable = true ) |-- _hoodie_partition_path: string (nullable = true ) |-- _hoodie_file_name: string (nullable = true ) |-- origin: string (nullable = true ) |-- partitionpath: string (nullable = true ) |-- image: struct (nullable = true ) | |-- origin: string (nullable = true ) | |-- height: integer (nullable = true ) | |-- width: integer (nullable = true ) | |-- nChannels: integer (nullable = true ) | |-- mode: integer (nullable = true ) | |-- data: binary (nullable = true )
すべてのただの書き込みAPIを試しただけだが、想定通り書き込み・読み込みできている。
更新 Hudiといえば、テーブルを更新可能であることも特徴のひとつなので、試しに更新をしてみる。
元は同じデータであるが、「0」の画像データを新しいデータとしてDataFrameを定義し、更新する。
意図的に、一部のデータだけ更新する。
1 2 3 4 5 6 7 8 9 10 scala> val updateImageDir = "Downloads/mnist_jpg/trainingSet/trainingSet/0" scala> val updateDf = spark.read.format("image" ).option("dropInvalid" , true ).load(home + "/" + updateImageDir) scala> val updateHudiDf = updateDf.select($"image.origin" , partitionPath($"image.origin" ) as "partitionpath" , $"*" ) scala> updateHudiDf.write.format("hudi" ). option(TABLE_NAME , tableName). option(PRECOMBINE_FIELD_OPT_KEY , "origin" ). option(RECORDKEY_FIELD_OPT_KEY , "origin" ). option(PARTITIONPATH_FIELD_OPT_KEY , "partitionpath" ). mode(Append ). save(basePath)
以下のように、「0」のデータが更新されていることがわかる。
1 2 3 4 5 6 7 8 9 10 11 12 $ ls -R /tmp/hudi_images/ /tmp/hudi_images/: 0 1 2 3 4 5 6 7 8 9 /tmp/hudi_images/0: 86c74288-e541-4a25-addf-0c00e17ef6bf-0_0-29-14074_20200517135104.parquet 86c74288-e541-4a25-addf-0c00e17ef6bf-0_0-60-27759_.parquet /tmp/hudi_images/1: 4912032f-3365-4852-b2ae-91ffb5ea9806-0_1-29-14075_20200517135104.parquet (snip)
つづいて、更新されたことを確かめるため、改めて読み込みDataFrameを定義し、
1 2 3 4 scala> val updatedHudiImageDf = spark. read. format("hudi" ). load(basePath + "/*" )
試しに、差分がわかるように「1」と「0」のデータをそれぞれ読んで見る。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 scala> updatedHudiImageDf.select($"_hoodie_commit_time" , $"_hoodie_commit_seqno" , $"_hoodie_partition_path" ).filter("partitionpath == 1" ).show(3 ) +-------------------+--------------------+----------------------+ |_hoodie_commit_time|_hoodie_commit_seqno|_hoodie_partition_path| +-------------------+--------------------+----------------------+ | 20200517135104 | 20200517135104 _1_1| 1 | | 20200517135104 | 20200517135104 _1_12| 1 | | 20200517135104 | 20200517135104 _1_45| 1 | +-------------------+--------------------+----------------------+ only showing top 3 rows scala> updatedHudiImageDf.select($"_hoodie_commit_time" , $"_hoodie_commit_seqno" , $"_hoodie_partition_path" ).filter("partitionpath == 0" ).show(3 ) +-------------------+--------------------+----------------------+ |_hoodie_commit_time|_hoodie_commit_seqno|_hoodie_partition_path| +-------------------+--------------------+----------------------+ | 20200517140006 |20200517140006 _0_...| 0 | | 20200517140006 |20200517140006 _0_...| 0 | | 20200517140006 |20200517140006 _0_...| 0 | +-------------------+--------------------+----------------------+ only showing top 3 rows
上記のように、更新されたことがわかる。最新の更新日時は、
2020/05/17 14:00:06UTC である。
ここで読み込まれたデータも、Delta
Lake同様に画像を元にしたベクトルデータとして扱える。 Pythonであれば
ndarray に変換して用いれば良い。
Parquetの内容を確認する parquet-mr の
parquet-tools を利用して、Parquetの内容を確認する。
なお、Hudi
v0.5.2におけるParquetのバージョンは以下の通り、1.10.1である。
pom.xml:84
1 <parquet.version > 1.10.1</parquet.version >
当該バージョンに対応するタグをチェックアウトし、予めparquet-toolsをパッケージしておくこと。
スキーマ 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 $ java -jar target/parquet-tools-1.10.1.jar schema /tmp/hudi_images/0/86c74288-e541-4a25-addf-0c00e17ef6bf-0_0-60-27759_20200517140006.parquet message hoodie.hudi_images.hudi_images_record { optional binary _hoodie_commit_time (UTF8); optional binary _hoodie_commit_seqno (UTF8); optional binary _hoodie_record_key (UTF8); optional binary _hoodie_partition_path (UTF8); optional binary _hoodie_file_name (UTF8); optional binary origin (UTF8); optional binary partitionpath (UTF8); optional group image { optional binary origin (UTF8); optional int32 height; optional int32 width; optional int32 nChannels; optional int32 mode; optional binary data; } }
Hudiが独自に付加したカラムが追加されていることが見て取れる。
メタデータ つづいて、メタデータを確認する。
ファイル名やクリエイタなど。
1 2 file: file:/tmp/hudi_images/0/86c74288-e541-4a25-addf-0c00e17ef6bf-0_0-60-27759_20200517140006.parquet creator: parquet-mr version 1.10.1 (build a89df8f9932b6ef6633d06069e50c9b7970bebd1)
ブルームフィルタはここでは省略
1 2 3 extra: org.apache.hudi.bloomfilter = /// (snip)
レコードキーやAvroスキーマ
1 2 3 4 extra: hoodie_min_record_key = file:///home/centos/Downloads/mnist_jpg/trainingSet/trainingSet/0/img_1.jpg extra: parquet.avro.schema = {"type":"record","name":"hudi_images_record","namespace":"hoodie.hudi_images","fields":[{"name":"_hoodie_commit_time","type":["null","string"],"doc":"","default":null},{"name":"_hoodie_commit_seqno","type":["null","string"],"doc":"","default":null},{"name":"_hoodie_record_key","type":["null","string"],"doc":"","default":null},{"name":"_hoodie_partition_path","type":["null","string"],"doc":"","default":null},{"name":"_hoodie_file_name","type":["null","string"],"doc":"","default":null},{"name":"origin","type":["string","null"]},{"name":"partitionpath","type":["string","null"]},{"name":"image","type":[{"type":"record","name":"image","namespace":"hoodie.hudi_images.hudi_images_record","fields":[{"name":"origin","type":["string","null"]},{"name":"height","type":["int","null"]},{"name":"width","type":["int","null"]},{"name":"nChannels","type":["int","null"]},{"name":"mode","type":["int","null"]},{"name":"data","type":["bytes","null"]}]},"null"]}]} extra: writer.model.name = avro extra: hoodie_max_record_key = file:///home/centos/Downloads/mnist_jpg/trainingSet/trainingSet/0/img_9996.jpg
上記の通り、Hudiでは parquet-avro
を利用し、Avro形式でParquet内のデータを保持する。
スキーマ詳細
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 file schema: hoodie.hudi_images.hudi_images_record -------------------------------------------------------------------------------- _hoodie_commit_time: OPTIONAL BINARY O:UTF8 R:0 D:1 _hoodie_commit_seqno: OPTIONAL BINARY O:UTF8 R:0 D:1 _hoodie_record_key: OPTIONAL BINARY O:UTF8 R:0 D:1 _hoodie_partition_path: OPTIONAL BINARY O:UTF8 R:0 D:1 _hoodie_file_name: OPTIONAL BINARY O:UTF8 R:0 D:1 origin: OPTIONAL BINARY O:UTF8 R:0 D:1 partitionpath: OPTIONAL BINARY O:UTF8 R:0 D:1 image: OPTIONAL F:6 .origin: OPTIONAL BINARY O:UTF8 R:0 D:2 .height: OPTIONAL INT32 R:0 D:2 .width: OPTIONAL INT32 R:0 D:2 .nChannels: OPTIONAL INT32 R:0 D:2 .mode: OPTIONAL INT32 R:0 D:2 .data: OPTIONAL BINARY R:0 D:2
Rowグループ
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 row group 1: RC:4132 TS:4397030 OFFSET:4 -------------------------------------------------------------------------------- _hoodie_commit_time: BINARY GZIP DO:0 FPO:4 SZ:165/127/0.77 VC:4132 ENC:RLE,PLAIN_DICTIONARY,BIT_PACKED ST:[min: 20200517140006, max: 20200517140006, num_nulls: 0] _hoodie_commit_seqno: BINARY GZIP DO:0 FPO:169 SZ:10497/107513/10.24 VC:4132 ENC:RLE,PLAIN,BIT_PACKED ST:[min: 20200517140006_0_42001, max: 20200517140006_0_46132, num_nulls: 0] _hoodie_record_key: BINARY GZIP DO:0 FPO:10666 SZ:16200/342036/21.11 VC:4132 ENC:RLE,PLAIN,BIT_PACKED ST:[min: file:///home/centos/Downloads/mnist_jpg/trainingSet/trainingSet/0/img_1.jpg, max: file:///home/centos/Downloads/mnist_jpg/trainingSet/trainingSet/0/img_9996.jpg, num_nulls: 0] _hoodie_partition_path: BINARY GZIP DO:0 FPO:26866 SZ:100/62/0.62 VC:4132 ENC:RLE,PLAIN_DICTIONARY,BIT_PACKED ST:[min: 0, max: 0, num_nulls: 0] _hoodie_file_name: BINARY GZIP DO:0 FPO:26966 SZ:448/419/0.94 VC:4132 ENC:RLE,PLAIN_DICTIONARY,BIT_PACKED ST:[min: 86c74288-e541-4a25-addf-0c00e17ef6bf-0_0-60-27759_20200517140006.parquet, max: 86c74288-e541-4a25-addf-0c00e17ef6bf-0_0-60-27759_20200517140006.parquet, num_nulls: 0] origin: BINARY GZIP DO:0 FPO:27414 SZ:16200/342036/21.11 VC:4132 ENC:RLE,PLAIN,BIT_PACKED ST:[min: file:///home/centos/Downloads/mnist_jpg/trainingSet/trainingSet/0/img_1.jpg, max: file:///home/centos/Downloads/mnist_jpg/trainingSet/trainingSet/0/img_9996.jpg, num_nulls: 0] partitionpath: BINARY GZIP DO:0 FPO:43614 SZ:100/62/0.62 VC:4132 ENC:RLE,PLAIN_DICTIONARY,BIT_PACKED ST:[min: 0, max: 0, num_nulls: 0] image: .origin: BINARY GZIP DO:0 FPO:43714 SZ:16200/342036/21.11 VC:4132 ENC:RLE,PLAIN,BIT_PACKED ST:[min: file:///home/centos/Downloads/mnist_jpg/trainingSet/trainingSet/0/img_1.jpg, max: file:///home/centos/Downloads/mnist_jpg/trainingSet/trainingSet/0/img_9996.jpg, num_nulls: 0] .height: INT32 GZIP DO:0 FPO:59914 SZ:111/73/0.66 VC:4132 ENC:RLE,PLAIN_DICTIONARY,BIT_PACKED ST:[min: 28, max: 28, num_nulls: 0] .width: INT32 GZIP DO:0 FPO:60025 SZ:111/73/0.66 VC:4132 ENC:RLE,PLAIN_DICTIONARY,BIT_PACKED ST:[min: 28, max: 28, num_nulls: 0] .nChannels: INT32 GZIP DO:0 FPO:60136 SZ:111/73/0.66 VC:4132 ENC:RLE,PLAIN_DICTIONARY,BIT_PACKED ST:[min: 1, max: 1, num_nulls: 0] .mode: INT32 GZIP DO:0 FPO:60247 SZ:111/73/0.66 VC:4132 ENC:RLE,PLAIN_DICTIONARY,BIT_PACKED ST:[min: 0, max: 0, num_nulls: 0] .data: BINARY GZIP DO:0 FPO:60358 SZ:1609341/3262447/2.03 VC:4132 ENC:RLE,PLAIN,BIT_PACKED ST:[min: 0x00 (snip)